DAVID (https://david.ncifcrf.gov/)
연구자가 많은 유전자 목록 뒤에 숨겨진 생물학적 의미를 이해할 수 있도록포괄적인 기능적 주석 도구 세트를 제공하는
웹 사이트
간단한 사용방법
1. Start Analysis 카테고리로 들어가 Upload탭의 Step 1.에 원하는 유전자 목록을 넣는다.
이때, 줄로 구분하여 유전자 목록을 넣어야한다.

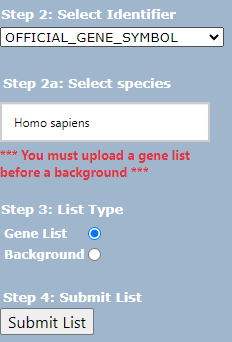
2. Step2의 select Identifier를 선택한다. 나는 주로 OFFICIAL_GENE_SYMBOL을 선택..!
이걸 선택하면 step 2a가 나타나는데, 나는 인간의 유전자를 분석하니까 homo sapiens를 넣어준다.(자동완성 기능 有)
그 후, step 3.에서 Gene List를 선택하여 Submit List를 누른다.

3. 그럼 페이지가 넘어가면서 List 탭이 선택되고, 내가 선택한 Homo sapiens와 List_1이 생성된 것을 볼 수 있다.
List_1이 내가 넣은 gene 목록, Rename으로 원하는 이름으로 바꿀 수 있다.
이름과 상관없이 분석을 보려면 오른쪽 Functional Annotation Tool을 눌러준다

4. 그럼 오른쪽 부분이 아래 사진과 같이 바뀐다. 각각을 눌러 원하는 주석 내용을 선택할 수 있다.
빨간색이 기본으로 선택되는 주석들..! 난 주로 기본에 REACTOME_PATHWAY만 더 선택해서 보곤한다..
그 후, Funtional Annotation Clustering 을 누른다.


5. 그럼 새 창으로 Annotation 결과가 나온다!
다운로드 파일을 눌러도 그냥 창에 글씨로 나와서.. 그냥 복사해서 csv파일로 만들고 있다.. ^^..

이 데이터들을 가져와서 p_value가 0.05 이하인 term들만을 확인..!
6. 만약 다른 gene list를 추가해서 보고싶다면, 원래 있던 화면에서 Upload 탭을 클릭해서 1부터 과정을 반복한다.
그럼 3번 과정에서와 다르게 List_2가 추가된 것을 확인할 수 있다. 원하는 목록을 선택하고 User를 누르면 끝!

인터넷 창을 끄지 않으면 데이터가 남아있는 것 같은데, 새 창을 키면 데이터가 사라지니 주의해야 한다.
이상 또 까먹기 싫어서 정리해 놓는 DAVID 분석 사용법 끝
댓글